تکنیک ها
توالی یابی ایلومینا چیست؟
توالی یابی ایلومینا تکنیکی است که برای تعیین ترتیب مجموعه ای از جفت بازها در DNA استفاده می شود که به عنوان یک روش توالی یابی DNA نیز شناخته می شود. مفهوم برگشت پذیری شیمیایی توسط برونو کانارد(Bruno Canard) و سیمون سرفاتی(Simon Sarfati) در موسسه پاستور در پاریس ابداع شد. توسط شانکاربالسوبرامانیان(Shankar Balasubramanian) ودیوید کلنرمان (David Klenerman) از دانشگاه کمبریج توسعه داده شد ، که متعاقبا شرکت Solexa را تأسیس کردند ، شرکتی که بعداً توسط کمپانی Illumina خریداری شد. این روش تعیین توالی بر اساس خاتمه دهنده های برگشت پذیر رنگی است که شناسایی نوکلئوتیدهای منفرد را هنگام شستشو روی رشته های DNA امکان پذیر می کند. همچنین می تواند برای تعیین توالی کل ژنوم و یا تعیین توالی یک ناحیه خاص ، تجزیه و تحلیل رونویسی ، متاژنومیکس ، کشف RNA کوچک ، و تجزیه و تحلیل برهمکنش پروتئین و نوکلئیک اسید در سطح ژنوم مورد استفاده قرار گیرد.
فناوری توالی Illumina در سه مرحله اساسی عمل می کند:
- افزایش(amplify)
- توالییابی (sequence)
- تجزیه و تحلیل(analyse)
این فرآیند با DNA خالص آغاز می شود. DNA تکه تکه شده و آداپتورهایی اضافه می شوند که شامل بخشهایی هستند که در حین مرحلههای مختلف تقویت ، تعیین توالی و تجزیه و تحلیل به عنوان نقاط مرجع عمل می کنند.DNA اصلاح شده بر روی یک فلوسل(flow cell) بارگذاری می شود که در آن تقویت و تعیین توالی انجام می شود. فلوسل حاوی نانولوله هایی است که قطعات را جدا کرده و شلوغی بیش از حد را کاهش میدهد. هر نانولوله حاوی الیگونوکلئوتیدها است که نقطه اتصال را برای اتصال آداپتورها فراهم می کند. پس از اتصال قطعات ، مرحله ای به نام تولید خوشه آغاز می شود. این مرحله حدود هزار نسخه از هر قطعه DNA ایجاد می کند و توسط PCR تقویت پل(bridge amplification PCR) انجام می شود. سپس ، پرایمرها و نوکلئوتیدهای اصلاح شده بر روی تراشه شسته می شوند. این نوکلئوتیدها دارای یک مسدود کننده فلورسنت
‘ 3 برگشت پذیر هستند بنابراین DNA پلیمراز می تواند فقط یک نوکلئوتید را در یک زمان به قطعه DNA اضافه کند. پس از هر دور سنتز ، یک دوربین از تراشه عکس می گیرد. یک کامپیوتر تعیین می کند که چه نوکلئوتیدی با توجه به طول موج برچسب فلورسنت(fluorescent tag)، اضافه شدهاست و آن را برای هر نقطه روی تراشه ثبت می کند. پس از هر دور ، مولکول های غیر ترکیب شده شسته می شوند. سپس یک مرحله رفع انسداد شیمیایی برای حذف گروه مسدود کننده ترمینال فلورسنت ‘3 استفاده می شود. این روند تا تعیین توالی کامل مولکول DNA ادامه می یابد. با استفاده از این فناوری ، هزاران مکان در سراسر ژنوم به طور همزمان از طریق توالی موازی عظیم توالی یابی می شوند.
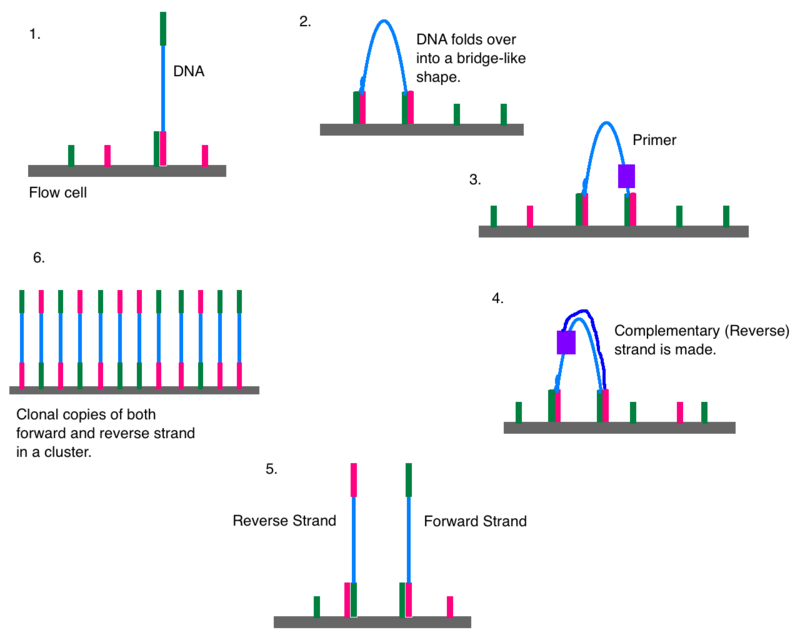
DNA از طریق توالی های مکمل به فلوسل متصل می شود. رشته خم می شود و به یک الیگو دوم متصل می شود و یک پل را تشکیل می دهد. پلیمراز رشته معکوس را سنتز می کند. دو رشته آزاد شده و راست می شوند. هر کدام یک پل جدید (تقویت پل) تشکیل می دهند. نتیجه این است که خوشه ای از کلون های رشته های رو به جلو و معکوس DNA ایجاد می شود.
کتابخانه ژنومیک
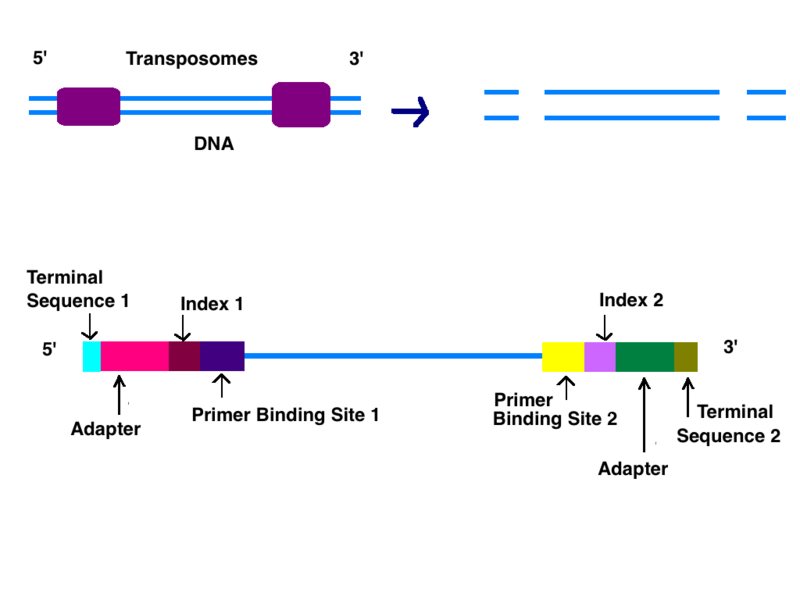
پس از خالص سازی DNA ، یک کتابخانه ژنومی ، باید تولید شود. به دو روش می توان کتابخانه ژنومی ایجاد کرد ، امواج فراصوت( sonification ) و (tagmentation) برچسب زدن .در tagmentation ترانسپوزازها به طور تصادفی DNA را به اندازه های بین 50 تا 500 قطعه bp برش می دهند و به طور همزمان آداپتورها را اضافه می کنند. همچنین می توان با استفاده از فراصوت برای خرد کردن DNA ژنومی ، یک کتابخانه ژنتیکی ایجاد کرد. در این روش با استفاده از امواج فراصوت DNA را به اندازه های مشابه تقسیم میشود. آداپتورهای راست و چپ باید پس از فراصوت توسط T7 DNA Polymerase و
T4 DNA ligase متصل شوند. رشته هایی که نمی توانند به آداپتورها متصل شوند، پاک می شوند.
DNA دو رشته ای توسط ترانسپوزوم ها شکافته می شود. انتهای برش ترمیم شده و آداپتورها ، شاخص ها ، محل اتصال پرایمر و محل های پایانی(ترمینال ها) به هر رشته DNA اضافه می شود.
Bridge amplification(افزایش پل)
پس از پیوستن ، تولید خوشه(cluster generation) می تواند آغاز شود. هدف این است که صدها رشته DNA یکسان ایجاد شود. برخی از آنها به عنوان رشته فوروارد (forward strand) خواهند بود. بقیه ، رشته معکوس (reverse). به همین دلیل است که از آداپتورهای راست و چپ استفاده می شود. خوشه ها از طریق تقویت پل ایجاد می شوند. DNA پلیمراز در امتداد یک رشته DNA حرکت می کند و رشته مکمل آن را ایجاد می کند. رشته اصلی شسته می شود و تنها رشته معکوس باقی می ماند. در بالای رشته معکوس یک توالی آداپتور وجود دارد. رشته DNA خم شده و به اولیگو متصل می شود که مکمل دنباله آداپتور بالا است. پلیمرازها به رشته معکوس متصل می شوند و رشته تکمیلی آن (که مشابه نسخه اصلی است) ساخته می شود. DNA دو رشته ای اکنون تغییر شکل می دهد به طوری که هر رشته می تواند جداگانه به یک توالی الیگونوکلئوتیدی متصل به فلوسل (flow cell) متصل شود. یکی رشته معکوس خواهد بود دیگری ، رشته فوروارد . این فرایند Bridge amplification (افزایش پل) نامیده می شود و برای هزاران خوشه در سراسر فلوسل به طور همزمان اتفاق می افتد.
میلیون ها الیگو در انتهای هر ردیف از فلوسل قرار دارند.
Clonal amplification
بارها و بارها ، رشته های DNA خم می شوند و به تکیه گاه جامد متصل می شوند. DNA پلیمراز یک رشته جدید را برای ایجاد یک بخش دو رشته ای سنتز می کند و آن را تغییر شکل می دهد به طوری که همه رشته های DNA در یک منطقه از یک منبع واحد (تقویت کلونال) هستند. تقویت کلونال برای اهداف کنترل کیفیت مهم است. اگر یک رشته دارای توالی فرد باشد ، دانشمندان می توانند رشته معکوس را بررسی کنند تا مطمئن شوند که مکمل همان توالی است. از آنجایی که توالییابی Illumina از DNA پلیمراز استفاده می کند ، خطاهای جایگزینی باز در این روش به ویژه در انتهای 3 ‘ مشاهده شده است ، . همخوانی انتهای زوج همراه با تولید خوشه می تواند تأیید کند که خطایی رخ داده است. رشته های reverse و forward باید مکمل یکدیگر باشند ، همه رشتههای معکوس و forward خوانده شده(توالی یابی شده) باید با یکدیگر مطابقت داشته باشند . اگر خواندهشده ها به اندازه کافی شبیه به همتایان خود (که باید با آن شبیه سازی شود) نباشد ، ممکن است خطایی رخ داده باشد. در تجزیه و تحلیل برخی آزمایشگاهها از حداقل آستانه 97٪ تشابه استفاده شده است.
تعیین توالی از طریق سنتز
در پایان مرحلهی قبل، همه رشته های reverse از فلوسل شسته می شوند و فقط رشته های forward باقی می مانند. پرایمر به محل اتصال پرایمر آداپتور رشته های forward متصل می شود و پلیمراز dNTP دارای برچسب فلورسنت را به رشته DNA اضافه می کند. فقط یک باز می تواند در هر دور اضافه شود زیرا فلوروفور به عنوان یک گروه مسدود کننده عمل می کند. با این حال ، گروه مسدود کننده برگشت پذیر است. با استفاده از تکنیک های شیمیایی ، هر یک از چهار باز دارای رنگ و طول موج منحصر به فرد هستند و پس از هر دور ، دستگاه ضبط می کند که کدام باز (چه طول موجی) اضافه شده است. پس از ثبت رنگ ، فلوروفور شسته می شود و dNTP دیگری روی flow cell شسته می شود و این روند تکرار می شود.
پس از خواندن رشته DNA ، رشته ای که تازه اضافه شده است شسته می شود. سپس ، پرایمر ایندکس 1 متصل می شود ، توالی ایندکس 1 را پلیمر می کند و شسته می شود. این رشته دوباره یک پل تشکیل می دهد و انتهای 3 ‘رشته DNA به یک الیگو در flow cell متصل می شود. پرایمر ایندکس 2 متصل می شود ، توالی را پلیمری می کند و شسته می شود.
یک پلیمراز رشته مکمل را در بالای رشته قوسی تعیین توالی می کند. آنها جدا می شوند و انتهای 3 ‘هر رشته مسدود می شود. رشته forward شسته می شود و روند دنباله توسط سنتز برای رشته معکوس تکرار می شود.
نوکلئوتیدهای برچسب زده شده به ترتیب به رشته DNA اضافه می شوند. هر یک از چهار نوکلئوتید دارای یک برچسب شناسایی هستند که می تواند برای انتشار طول موج مشخص تحریک شود. یک کامپیوتر همه انتشارات را ثبت می کند و از این داده ها ، توالی و ترتیب باز ها مشخص می شود.
تحلیل داده ها
تعیین توالی برای میلیون ها خوشه به طور همزمان اتفاق می افتد و هر خوشه دارای 1000 کپی یکسان از DNA وارد شده به سیستم است. داده های توالی با یافتن قطعاتی با مناطق همپوشان ، که کانتیگ(contigs) نامیده می شوند ، تجزیه و تحلیل می شوند. اگر توالی مرجع شناخته شده داشته باشد ، کانتیگ ها برای شناسایی با آن مقایسه می شوند.
این فرایند قطعه قطعه به دانشمندان اجازه می دهد تا توالی کامل را ببینند حتی اگر یک توالی بدون قطعه هرگز اجرا نشده باشد. اگر دنباله به صورت نو باشد و مرجعی وجود نداشته باشد ، مناطق تکراری می توانند در مونتاژ دنباله مشکلات زیادی ایجاد کنند. مشکلاتی مانند جایگزینی های بازها (به ویژه در انتهای’ 3) توسط پلیمرازهای نادرست ، توالی کیمریک و PCR-bias است ، که همه آنها می توانند به ایجاد توالی نادرست کمک کنند.
مترجم: آتوسا بهنام راد




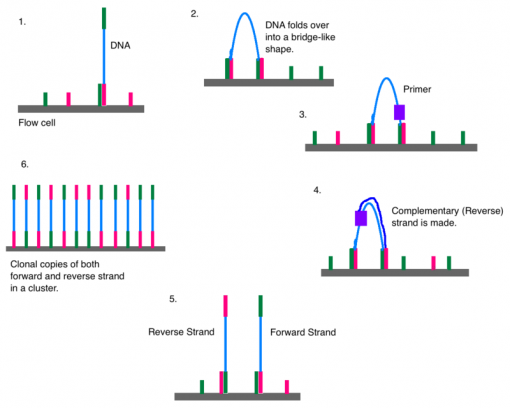
بویژه قسمت مربوط به خوانش توالی های ایندکس ۱ و ۲.
سلام،
عالی بود.