بیوانفورماتیک, تکنیک ها
طراحی پرایمر چیست؟
طراحی پرایمر یکی از مباحث فوق العاده با اهمیت در مباحث مولکولی می باشد. چرا که تا حد زیادی موفقیت تکنیک PCR و Real-Time PCR در گرو طراحی پرایمر مناسب می باشد. طراحی پرایمر نامناسب می تواند منجر به عدم تکثیر قطعه مورد نظر در PCR، تولید کم محصول PCR و یا از سوی دیگر منجر به تولید غیر اختصاصی محصولات PCR می گردد.
لذا در طراحی پرایمر بایستی دقت کافی اعمال شود. برای طراحی پرایمر ابتدا بایستی توالی ژن مورد مطالعه را به فرمت FASTA از بانک های توالی اسیدنوکلئیک بدست آورد. یکی از معروف ترین بانک های توالی نوکلئوتیدی سایت NCBI می باشد.
در مرحله ی بعدی از راه های مختلفی می توان اقدام به طراحی پرایمر نمود. یکی از نرم افزار های کاربردی به منظور طراحی پرایمر، نرم افزار gene runner می باشد. در طراحی پرایمر بایستی به یکسری پارامترهایی از جمله طول پرایمر، درصد GC، ساختارهای runs، دمای Tm و دمای annealing، عدم وجود ساختارهای ثانویه مانند دایمر پرایمر، GC clamp و اختصاصی بودن پرایمر طراحی شده، توجه نمود. در اینجا به اختصار به آموزش طراحی پرایمر به زبان ساده می پردازیم.
طول پرایمر بایستی بین 18 تا 24 نوکلئوتید در نظر گرفته شود. پرایمرهای با طول کمتر از 18 نوکلئوتید به دلیل احتمال شباهت زیاد به مکان های متعددی از ژنوم متصل شده و در نتیجه منجر به تولید محصولات غیر اختصاصی در PCR می گردد.
در مورد پارامتر درصد GC، بهترین محدوده ی توصیه شده بین 40 تا 60 می باشد. اختلاف درصد GC بین پرایمرهای رفت و برگشت نبایستی بیشتر از 5 درصد باشد.
در مورد معیار دمای Tm، بهترین محدوه ی توصیه شده بین 50 تا 60 درجه می باشد. اختلاف دمای Tm بین پرایمرهای رفت و برگشت توصیه می شود بیشتر از 2 درجه نباشد.
در طراحی پرایمر بایستی از وجود ساختار های runs( توالی های تکراری پشت سر هم) و وجود ساختارهای ثانویه مانند ساختار های ساقه-حلقه (stem loop)، ساختار های سنجاق سری (hairpin) و ساختارهای دایمر پرایمر اجتناب نماییم.
از آنجایی که سر ‘3 پرایمر از اهمیت خاصی در تکثیر قطعه ی مورد مطالعه برخوردار است بایستی از اتصال مناسب آن اطمینان حاصل نماییم که بهتر است پرایمر در سر ‘3 خود دارای C و یا G باشد.
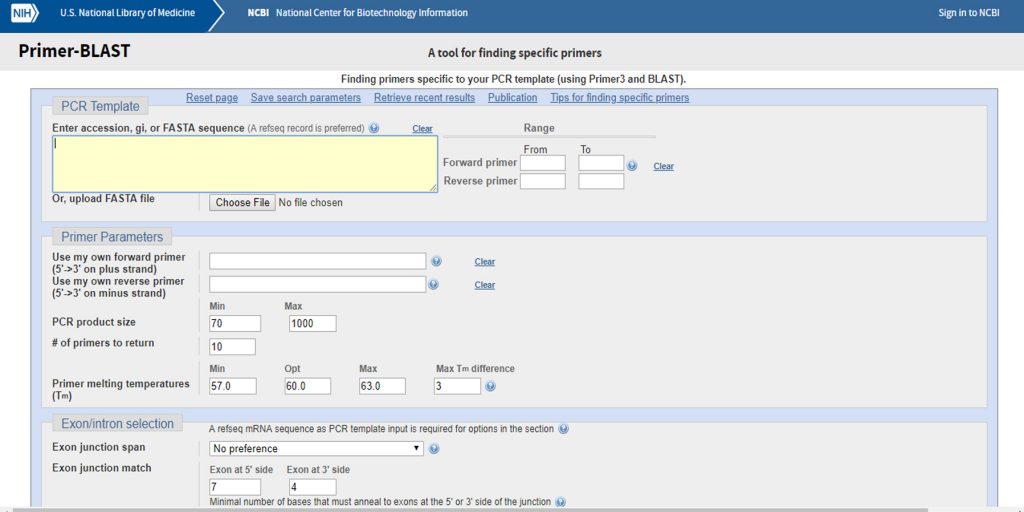
اتصال اختصاصی پرایمر را می توان از طریق سایت Primer Blast آنالیز نمود.




سلام وقت بخیر. برای ژنهایی که کلا splicing ندارن چطوری پرایمر طراحی میشود؟
با سلام برای ژن های تگ اگزونه و ژن های موجودات پروکاریوتی می بایست از آنزیم DNase به منظور حذف آلودگی DNA ژنومیک استفاده کرد
سلام من در سايت ncbi بعد از انتخاب گزينه GENE ،و وارد كردن اسم ژن موردنظر،در قسمت Gene table بايد طبق گفته استادم بروم و اگزون هاى مشترك را در نظر بگيرم و توالى ان ها را در بلست بزنم
حالا سوال من اين هست كه در اگزون مشترك ،تك تك اگزون ها رو توالى شان را بايد در بلست بزنم يا ٢ تا اگزون را با هم ،تواليشان را بردارم و در بلاست بزنم ؟
سوال بعديم اينه كه در اليگو پرايمر مدنظرى كه در ncbi به دست اوردم ،تواليش را در اليگو زدم كه ببينم ايا خوب هست يا نه؟ پرايمر reverse و Forward كه ميده مثل اونى كه در ncbi به دست اوردم ،نيست و دلتاش هم خيلي منفى و پرايمر نامناسبيه،در صورتى كه در ncbi به نظر از بقيه بهتره
نمى دونم منظورم را تونستم به درستى بيان كنم ؟!
ممنون ميشم پاسخ دهيد
سلام همه توالی را باید در بلاست قرار دهید.
سلام
من میخوام برای یک ژن پرایمر طراحی کنم.
سوال من اینه که آیا در نقاط اینترونی و غیر فعال ژن میتوان پرایمر فوروارد و ریورس طراحی کرد؟
سلام بله
سلام خسته نباشیدچجوری طراحی پرایمر و بیو انفورماتیک یاد بگیریم
سلام دوست عزیز
برای طراحی پرایمر یک کارگاه داریم که فعلا مشخص نیست چه زمانی برگزار میشود اما این تکنیک در دوره کارآموزی ژنتیک مولکولی آموزش داده خواهد شد. همچنین درباره بیوانفورماتیک میتوانید با آزمایشگاه تماس بگیرید تا مشاوره رایگان دریافت کنید. بیوانفورماتیک نیز یک دوره جداگانه دارد. لطفا برای اطلاعات بیشتر هر دوره روی هر لینک زیر کلیک کنید:
کارآموزی بیوانفورماتیک
کارآموزی ژنتیک مولکولی
سلام لطفا در مورد مرحله اول طراحی پرایمر قبل از وارد کردن توالی در الیگو، یعنی پیدا کردن توالی ژن مد نظر از سایت ncbi راهنماییم کنید. ممنون.
سلام لطفا NCBI رو سرچ کنید و وارد پایگاه بشید در قسمت بالای فیلد باید گزینه GENE رو انتخاب کنید . اسم ژن مورد نطرتون رو تایپ کرده ودر پایگاه سرچ کنید. سپس یک صفحه براتون باز میشه که جدول داره و همه ی ژن ها و زیرمجموعه های RNA اون ژن و انواع اون ژن در سایر موجودات ازمایشگاهی رو داراست . با توجه به فیلد مورد نظرتون) مثلا گزینه هوموساپینس رو انتخاب کنید.ژن رو با کلیک باز کرده و در صفحه بعدباید اسکرول داون کرده و به پایین صفحه تشریف ببرید و یک قسمت داره به نام NCBI Reference Sequences (RefSeq) از این قسمت گزینه Download
GenBank, FASTA, Sequence Viewer (Graphics) را انختاب کرده( فستا) و وارد صفحه بعد بشیدد.(البته با توجه به میزان طول و لوکیشنی که از ژن مد نظرتون میخواید میتونید مشخصات طول رو خونده و بعد از این قسمت رفرنس سیکونس ژن رو انتخاب کنید)
سپس توالی را یا میتونید کپی پیست کرده و داخل ورد بندازید یا از گزینه sEND TO ،بعد FILE و بعد FASTA را دانلود کنید.