بیوانفورماتیک، تکنیک ها، ویدیو های آموزشی
ابزارها و نرم افزارهای آنالیز شبکه و مسیر (Panther, Reactome, KEGG, Cytoscape,…)
آنالیز شبکه و مسیرهای بیولوژیکی
آنالیز مسیر Pathway analysis غالبا به منظور gene enrichment یا غنی سازی ژن انجام می شود که در نتیجه ی آن می توان به مکانیسم های بیولوژیکی ایجاد شده توسط ژن مورد نظر و همچنین اطلاعات عملکردی ژن مورد نظر پی برد. آنالیز مسیر یا pathway analysis در کنار بررسی مسیرهای بیولوژیکی ژن ها، روابط بین ژن های مورد مطالعه را نیز بررسی می کند.
با پیشرفت علم اومیکس در تمامی گرایش های آن از جمله ژنومیکس، ترانسکریپتومیکس، پروتئومیکس و متابولومیکس، دسترسی به داده های اومیکس در تمامی موارد اشاره شده وجود دارد و در نتیجه برهمکنش بین ژن ها در مسیرهای بیولوژیکی، و همچنین روابط بین ژن ها و محصولاتشان در سطوح مختلفی RNA و پروتئین آشکار شده است.
آنالیز مسیرهای بیولوژیکی یکی از مباحث مهم و کاربردی در علم بیوانفورماتیک می باشد که برای این منظور ابزارهای بیوانفورماتیکی و همچنین دیتابیس های بیوانفورماتیکی تهیه شده است که از جمله مهم ترین آنها می توان به KEGG، Reactome، Panther، BioCarta، WikiPathways، NCI و GO اشاره نمود.
نگاهی از ابزارها و نرم افزارهای آنالیز شبکه و مسیر
پایگاه داده Reactome

Reactome از جمله پایگاه داده ها در زمینه ی آنالیز مسیرهای بیولوژیکی است که هم open source و هم open access می باشد. هدف پایگاه داده ی Reactome ارائه ی ابزارهای بیوانفورماتیکی بصری به منظور تجسم، تفسیر و تجزیه و تحلیل مسیرهای بیولوژیکی برای حمایت از تحقیقات پایه و بالینی، تجزیه و تحلیل ژنوم، مدل سازی و تحقیقات مربوط به سیستم بیولوژی می باشد. پروژه ی Reactome در سال 2003 تاسیس شد و توسط لینکلن استاین از OICR، پیتر دی استاشیو از NYULMC، هنینگ هرمجاکوب از EMBL-EBI و گوانمینگ وو از OHSU رهبری و کنترل می شود.
همانطور که گفته شد پایگاه داده ی Reactome یک پایگاه داده ی open source در زمینه ی مولکول های سیگنالینگ و متابولیک و روابط آنها در مسیرها و فرآیندهای بیولوژیکی است. واحد اصلی مدل داده Reactome واکنش یا reaction است. اسیدهای نوکلئیک، پروتئینها، کمپلکسها، واکسنها، داروهای ضد سرطان و مولکولهای کوچک که در واکنشها شرکت میکنند شبکهای از فعل و انفعالات بیولوژیکی را تشکیل میدهند و در مسیرهای مختلفی گروهبندی میشوند. نمونه هایی از مسیرهای بیولوژیکی در Reactome شامل متابولیسم واسطه کلاسیک، سیگنالینگ، تنظیم رونویسی، آپوپتوز و بیماری است.
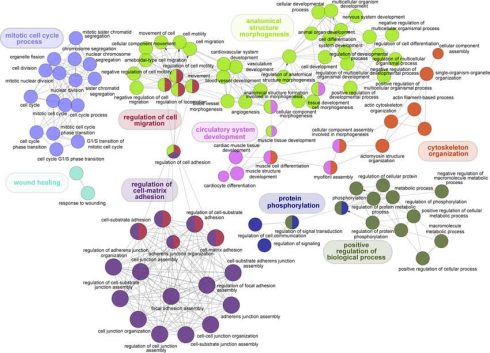
پایگاه داده ی Reactome طوری طراحی شده است که به معنای واقعی کلمه یک نقشه گرافیکی از فرآیندها و مسیرهای بیولوژیکی شناخته شده را به کاربر ارائه می دهد که همچنین رابطی است که کاربر می تواند برای اطلاعات دقیق در مورد اجزا و روابط آنها استفاده کند. پایگاه داده و وب سایت Reactome دانشمندان، محققان، دانش آموزان و مربیان را قادر می سازد تا اطلاعات بیولوژیکی را برای پشتیبانی از تجسم، ادغام و تجزیه و تحلیل داده ها پیدا کنند و به درستی سازماندهی و استفاده نمایند.
صفحات مربوط به مسیرها، واکنش و مولکول ها در پایگاه داده ی Reactome به طور گسترده به بیش از 100 منبع آنلاین مختلف بیوانفورماتیکی، از جمله پایگاه های داده NCBI Gene، Ensembl و UniProt، مرورگر ژنوم UCSC ، پایگاه داده های مولکول های کوچک ChEBI و پایگاه داده های PubMed ارجاع داده می شوند.
پایگاه داده ی Reactome توسط پزشکان، متخصصان ژنتیک، محققان ژنومیک و زیست شناسان مولکولی برای تفسیر نتایج مطالعات تجربی high throughput، توسط متخصصان بیوانفورماتیکی که به دنبال توسعه الگوریتم های جدید برای استخراج دانش از مطالعات ژنومی هستند و همچنین توسط متخصصان سیستم بیولوژی که مدل های پیش بینی در شرایط نرمال و شرایط بیماری را ایجاد می کنند، استفاده می شود.
تمامی داده ها و نرم افزارها به صورت رایگان برای دانلود در دسترس هستند. دادههای مربوط به واکنش ها، مسیر ها و تعاملات به صورت فایلهای Flat، Neo4j GraphDB، MySQL، BioPAX، SBML و PSI-MITAB قابل دانلود ارائه میشوند و از طریق APIهای خدمات وب سایت Reactome قابل دسترسی هستند. نرمافزار و دستورالعملهای نصب محلی پایگاهداده، وبسایت و ابزارهای ورود داده Reactome نیز از طریق سایت Reactome به آدرس https://reactome.org/ در دسترس می باشند.
نرم افزار Cytoscape

نرم افزار cytoscape یک نرم افزار open source می باشد که برای تجزیه و تحلیل شبکه ها و تجسم نمودار می توان از آن استفاده نمود. نرم افزار Cytoscape به کاربر این امکان را می دهد که براحتی نمودارهای تعاملی را نمایش داده و دستکاری نماید. نرم افزار cytoscape یک نرم افزار کاربردی در علم بیوانفورماتیک به منظور ترسیم و تحلیل شبکه های ژنی و آنالیز مسیر یا Pathway analysis می باشد.
از ویژگی های اصلی نرم افزار Cytoscape می توان به دارا بودن کتابخانه جاوا اسکریپت و کامپونتی به نام Cytoscape.js اشاره نمود که کتابخانه ی java script برای آنالیز و تجزیه و تحلیل شبکه بکار می رود و Cytoscape.js به منظور استفاده در برنامه های تحت وب می باشد.
از جمله ویژگی های منحصر بفرد نرم افزار Cytoscape می توان به وجود پلاگین (Plugins) اشاره کرد که این قابلیت را به نرم افزار Cytoscape می دهد که کاربردهای جدید از طریق این پلاگین یا افزونه به نرم افزار cytoscape اضافه گردد. برای اطلاعات بیشتر در مورد نرم افزار cytoscape می توانید به سایت https://js.cytoscape.org و همچنین سایت GitHub README مراجعه کنید.
یکی از کاربردهای اصلی نرم افزار cytoscape در علم سیستم بیولوژی یا زیست شناسی سامانه ای می باشد که امروز به عنوان بخش مهمی از فعالیت های بیوانفورماتیکی شناخته می شود. علم سیستم بیولوژی به مطالعه ی ارتباط بین اجزاء بیولوژیکی می پردازد. سیستم های بیولوژیکی ماهیتی زنده و پیچیده دارند فلذا پیش بینی و بررسی رفتار تک تک اجزا مشکل است.
علم سیستم بیولوژی با نگاهی سیستماتیک و با استفاده از ابزارهای بیوانفورماتیکی و بهره گیری از علم اومیکس در تمامی شاخه های آن (ژنومیکس، ترنسکریپتومیکسف پروتئومیکس و متابولومیکس) رفتار دینامیکی کل سیستم را پیشبینی و آنالیز می کند. علم زیست شناسی سامانه ای یا سیستم بیولوژی با دید وسیع و کلی خود به سیستم های زیستی به محققان کمک می کند تا درک بهتری از اینترکشن های اجزاء یک سیستم بیولوژیکی مانند DNA، RNA، پروتئین و متابولیت ها در شرایط و زمان های مختلف داشته باشند.
پایگاه داده ی PANTHER
سیستم طبقهبندی PANTHER بر مبنای تجزیه و تحلیل پروتئین از طریق روابط تکاملی می باشد که برای طبقهبندی پروتئینها و ژنهای آنها به منظور تسهیل تجزیه و تحلیل با توان بالا طراحی شده است. هسته PANTHER یک کتابخانه جامع و مشروح از درختان فیلوژنتیک خانواده ژنی است. همه گرههای درخت دارای شناسههای دائمی هستند که بین نسخههای PANTHER نگهداری میشوند و بستری پایدار برای حاشیهنویسی یا annotation ویژگیهای پروتئین مانند زیرخانواده و عملکرد، فراهم میکنند.
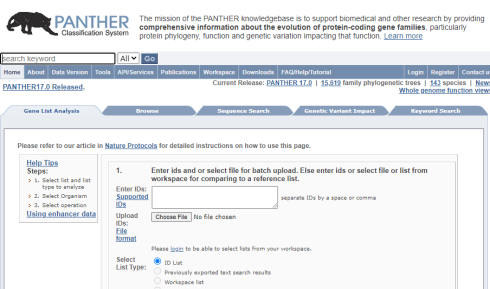
هر درخت فیلوژنتیک برای حاشیه نویسی یا annotation هر یک از اعضای پروتئینی خانواده با موارد زیر استفاده می شود:
- خانواده و کلاس پروتئین (سوپرگروه خانواده های پروتئینی)
- زیرخانواده (زیرگروه در درخت های فیلوژنتیک خانواده)
- ارتولوگ ها (ژن های موجود در سایر موجودات که از همان ژن در MRCA مشتق می شوند)
- پارالوگ ها (ژن های موجود در همان ارگانیسم که با تکرار ژن به هم مرتبط هستند)
- تابع (با استفاده از اصطلاح GO حاشیه نویسی شده بر روی درختان)
- مسیرها ( با سرپرستی PANTHER و Reactome)
اطلاعات طبقه بندی شده ی ژن کد کننده پروتئین را می توان در وب سایت PANTHER جستجو کرد یا برای ژن های ۱۴۳ ژنوم موجود در درختان PANTHER دانلود کرد. برای طبقهبندی ژنهایی که در درختها نیستند، توصیه میکنیم ابزار TreeGrafter را دانلود کرده و آن را روی رایانه شخصی خود اجرا کنید.
طبقهبندیهای PANTHER نتیجه حمایت های انسانی و همچنین الگوریتمهای پیچیده بیوانفورماتیک است.
برای اطلاعات بیشتر در مورد داده های PANTHER و روابط بین انواع داده ها، به شکل “PANTHER Data Overview” مراجعه کنید. خانوادهها و درختهای PANTHER سالانه مجددا محاسبه میشوند تا بهروزرسانیها در خانواده PANTHER و همچنین در فهرست ژنهای کدکننده پروتئین در هر ژنوم و توالیهای پروتئینی آنها منعکس شوند، همانطور که در UniProt Reference Proteomes نشان داده شده است.
PANTHER توسط بنیاد ملی علوم پشتیبانی می شود و توسط آزمایشگاه توماس در دانشگاه کالیفرنیای جنوبی نگهداری می شود. نسخه های قبلی PANTHER توسط Celera Genomics و کمک های مالی تحقیقاتی از موسسه ملی علوم پزشکی عمومی و بنیاد ملی علوم پشتیبانی شده است.
نسخه 17.0 PANTHER که در سال 22-02-2022 منتشر شد شامل ۱۵۶۱۹ خانواده پروتئینی است که به ۱۲۴۶۳۲ زیرخانواده پروتئینی متمایز از نظر عملکردی تقسیم می شوند.
پایگاه داده ی KEGG
KEGG (Kyoto Encyclopedia of Genes and Genomes) یک پایگاه داده برای تجزیه و تحلیل سیستماتیک عملکردهای ژن است که اطلاعات ژنومی را با اطلاعات عملکردی مرتبط می کند.
از سال ۱۹۹۵ پایگاه داده ی KEGG در آزمایشگاه Kanehisa در دانشگاه Kyoto و همچنین دانشگاه توکیو با استفاده از بودجه ی وزارت آموزش و آژانس های آن توسعه یافته است. برخلاف تصور عمومی، KEGG هرگز یک پایگاه داده عمومی نبوده است زیرا هرگز تعهد بلندمدت رسمی از هیچ سازمان دولتی وجود نداشته است.
KEGG در حال حاضر یکی از پرکاربردترین پایگاه های بیولوژیکی در جهان است که توسط آمار دسترسی به وب، ۱۵۰ تا ۲۰۰ هزار بازدیدکننده منحصر به فرد در ماه دارد و تعداد استنادات مقاله KEGG یک هزار در سال گزارش شده است.
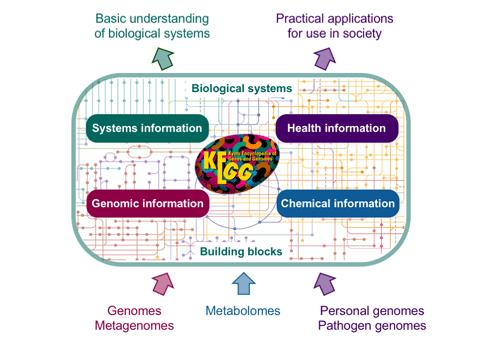
اطلاعات ژنومی در پایگاه داده GENES ذخیره میشود، که مجموعهای از کاتالوگهای ژنی برای همه ژنومهای کاملا توالی یابی شده و برخی ژنومهای جزئی با حاشیهنویسی بهروز از عملکردهای ژن است. اطلاعات عملکردی در پایگاه داده PATHWAY ذخیره می شود، که حاوی نمایش های گرافیکی فرآیندهای سلولی، مانند متابولیسم، انتقال غشاء، انتقال سیگنال و چرخه سلولی است.
پایگاه داده PATHWAY با مجموعه ای از جداول گروه ارتولوگ برای اطلاعات مربوط به مسیرهای فرعی حفاظت شده تکمیل می شود، که اغلب توسط ژن های جفت موقعیتی روی کروموزوم کدگذاری می شوند و به ویژه در پیش بینی عملکرد ژن مفید هستند. سومین پایگاه داده در KEGG LIGAND برای اطلاعات مربوط به ترکیبات شیمیایی، مولکول های آنزیم و واکنش های آنزیمی است.
KEGG ابزارهای گرافیکی جاوا را برای مرور نقشههای ژنوم، مقایسه دو نقشه ژنوم و دستکاری نقشههای بیان و همچنین ابزارهای محاسباتی برای مقایسه توالی، مقایسه نمودار و محاسبه مسیر ارائه میکند. پایگاه داده های KEGG روزانه به روز می شوند و به صورت رایگان در دسترس هستند که می توان از لینک (http://www.genome.ad.jp/kegg/) استفاده نمود.
از صفحات زیر بازدید کنید:




